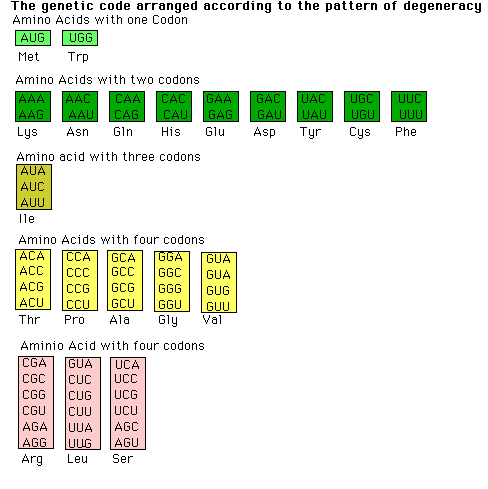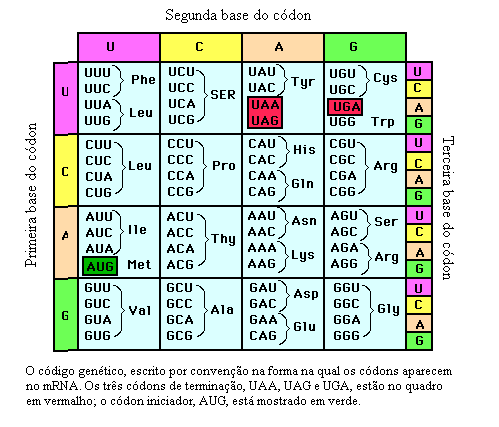

Algumas moléculas de tRNA podem reconhecer mais de um códon devido à oscilação no pareamento de bases. Por exemplo, o tRNA de alanina de levedura liga-se a três códons; GCC, GCU e GCA. As primeiras duas bases desses códons são iguais, enquanto a terceira é diferente.
Podem ser feitas duas generalizações quanto à interação códon-anticódon:
1. As primeiras duas bases de um códon pareiam-se do modo-padrão. O reconhecimento é exato. Os códons que diferem por suas duas primeiras bases devem ser reconhecidos por diferentes tRNAs.
2. A primeira base de um anticódon define se uma determinada molécula de
tRNA lê um, dois ou três tipos de códon: C ou A (um códon), U ou G (dois códons),
ou I (três códons). Assim, parte da redundância do código genético surge da imprecisão
(oscilação) no pareamento da terceira base do códon com a primeira base do anticódon.
Vemos aqui um forte motivo para o freqüente aparecimento de inosina, um dos nucleotídeos
incomuns nos anticódons. A inosina maximiza o número de códons que podem ser lidos por
uma determinada molécula de tRNA. As inosinas no tRNA são formadas por desaminação
de adenosina após a síntese do transcrito primário.